资讯专栏INFORMATION COLUMN

此篇文章关键给大家介绍了Python完成一阶矩马尔可夫过程形成任意DNA序列实例详细说明,感兴趣的小伙伴可以参考借鉴一下,希望可以有一定的帮助,祝愿大家多多的发展,尽早涨薪。
1.基本原理
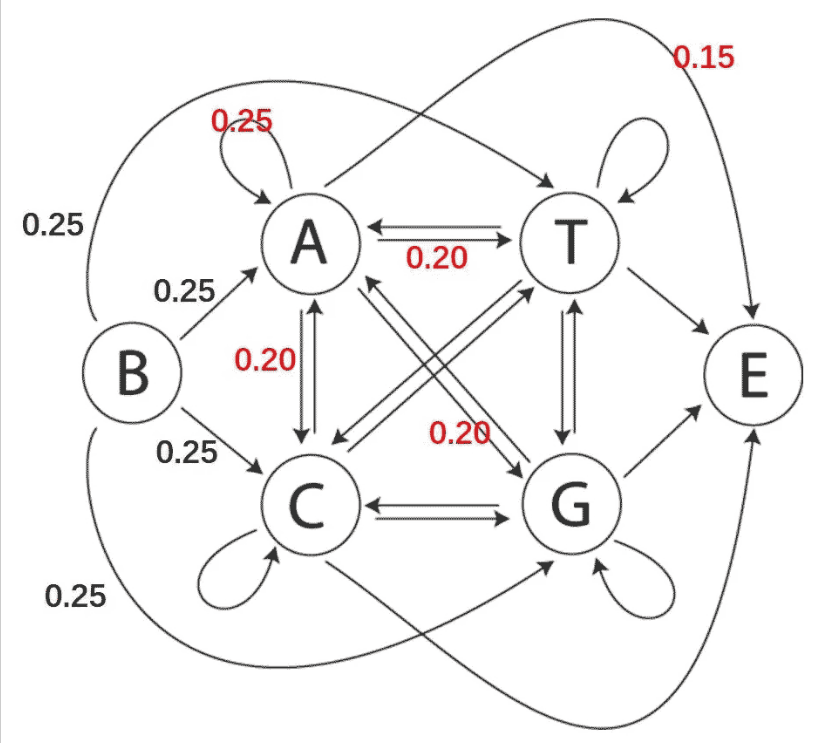
针对DNA序列,一阶矩马尔可夫过程可以看作现阶段碱基对的种类仅在于上一位碱基对种类。如下图1所示,1条编码序列的开始(由B逐渐)有可能是A、T、G、C4种碱基对(且概率同样,均是0.25),若编码序列某还有一位A,则下一个碱基对是A、T、G、C的几率依次为0.25、0.20、0.20、0.20,下一个无碱基对(即编码序列完毕,情况为E)的几率为0.15。
图1 DNA序列的一阶马尔科夫链
2.代码实现
以下代码运行于Jupyter Notebook(Python 3.7);代码功能是随机生成一定数量的DNA序列,统计序列长度并绘制分布图。若希望显示随机生成的序列,将代码#print(''.join(Seq))前的#删除即可。
import numpy
import random
import seaborn as sns
import matplotlib.pyplot as plt
#状态空间
states=["A","G","C","T","E"]
#可能的事件序列
transitionName=[["AA","AG","AC","AT","AE"],
["GA","GG","GC","GT","GE"],
["CA","CG","CC","CT","CE"],
["TA","TG","TC","TT","TE"],]
#概率矩阵(转移矩阵)
transitionMatrix=[[0.25,0.20,0.20,0.20,0.15],
[0.20,0.25,0.20,0.20,0.15],
[0.20,0.20,0.25,0.20,0.15],
[0.20,0.20,0.20,0.25,0.15]]
def RandomDNAs(Num):
max_len=0
i=0
Seq=[]#创建列表(Seq)用于添加碱基,以组成DNA序列
Len=[]#创建列表(Len)用于记录每条生成序列的长度
while i!=Num:
Base=["A","G","C","T"]
START=random.choice(Base)#随机从碱基中选择一个作为序列的起始碱基
Seq.append(START)#将起始碱基添加至Seq中
while START!="E":
if START=="A":
change=numpy.random.choice(transitionName[0],p=transitionMatrix[0])
#以transitionMatrix矩阵第一行的概率分布随机抽取transitionName第一行包含的事件
if change=="AA":
START="A"#如果转移状态是AA(即A碱基接下来的碱基是A,则将起始碱基设为A)
elif change=="AG":
START="G"
elif change=="AC":
START="C"
elif change=="AT":
START="T"
elif change=="AE":
START="E"
elif START=="G":
change=numpy.random.choice(transitionName[1],p=transitionMatrix[1])
if change=="GA":
START="A"
elif change=="GG":
START="G"
elif change=="GC":
START="C"
elif change=="GT":
START="T"
elif change=="GE":
START="E"
elif START=="C":
change=numpy.random.choice(transitionName[2],p=transitionMatrix[2])
if change=="CA":
START="A"
elif change=="CG":
START="G"
elif change=="CC":
START="C"
elif change=="CT":
START="T"
elif change=="CE":
START="E"
elif START=="T":
change=numpy.random.choice(transitionName[3],p=transitionMatrix[3])
if change=="TA":
START="A"
elif change=="TG":
START="G"
elif change=="TC":
START="C"
elif change=="TT":
START="T"
elif change=="TE":
START="E"
if START!="E":
Seq.append(START)#如果状态转移后不为End(E),则将转移后的碱基加到Seq序列中
i+=1
Len.append(len(Seq))
if len(Seq)>max_len:
max_len=len(Seq)
#print(''.join(Seq))
Seq.clear()
plt.hist(numpy.array(Len),bins=max_len,edgecolor="white")
#显示横轴标签
plt.xlabel("DNA Sequence Length")
#显示纵轴标签
plt.ylabel("Frequency")
#显示图标题
plt.title("Histogram of frequency distribution of DNA sequence length")
plt.show()
print("DNA序列的最大长度为:",max_len)
print("DNA序列长度的众数为:",max(Len,key=Len.count))
%matplotlib notebook#若未使用Jupyter Notebook,此句不需要
RandomDNAs(1000)#1000表示随机生成1000条序列3.运行结果
从以下4个序列长度分布统计可以看到,随着随机生成的序列数量增多,序列长度分布愈发集中,且长度为1bp的序列占比最多且逐渐增加。
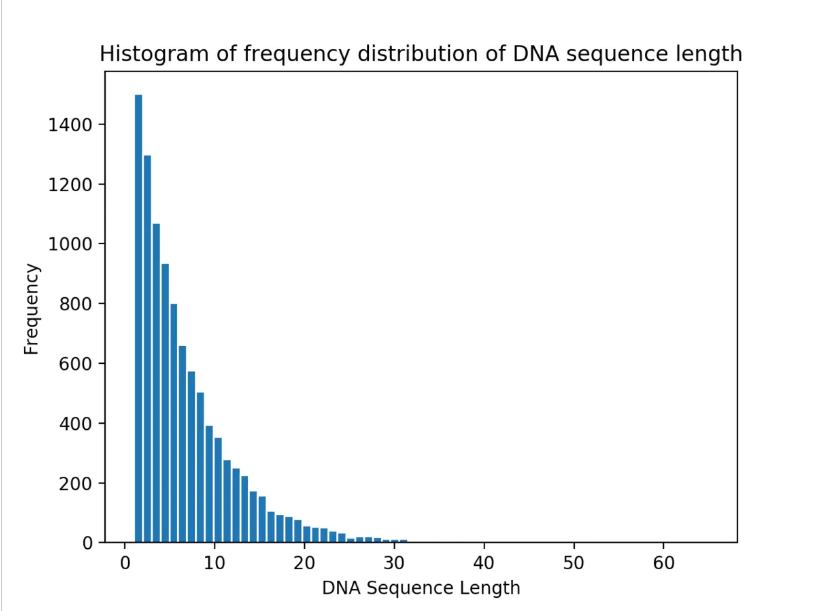
图2 10,000条DNA序列的序列长度分布统计
10,000条DNA序列的序列中
DNA序列的最大长度为:65
DNA序列长度的众数为:1
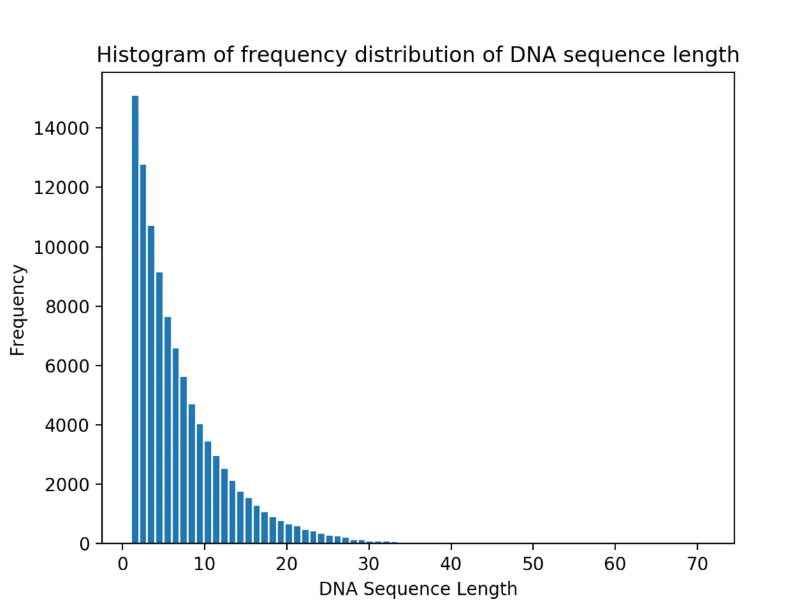
图3 100,000条DNA序列的序列长度分布统计
100,000条DNA序列的序列中
DNA序列的最大长度为:71
DNA序列长度的众数为:1
综上所述,这篇文章就给大家介绍到这里了,希望可以给大家带来帮助。
文章版权归作者所有,未经允许请勿转载,若此文章存在违规行为,您可以联系管理员删除。
转载请注明本文地址:https://www.ucloud.cn/yun/128699.html
摘要:本文将讨论两种可用于解决贝叶斯推理问题的主要方法基于采样的马尔可夫链蒙特卡罗,简称方法和基于近似的变分推理,简称方法。而贝叶斯推理则是从贝叶斯的角度产生统计推断的过程。贝叶斯推理问题还可能会产生一些其他的计算困难。 全文共6415字,预计学习时长20分钟或更长 showImg(https://segmentfault.com/img/bVbvFZZ?w=1280&h=853); 图片来...
摘要:近来在深度学习中,卷积神经网络和循环神经网络等深度模型在各种复杂的任务中表现十分优秀。机器学习中最常用的正则化方法是对权重施加范数约束。 近来在深度学习中,卷积神经网络和循环神经网络等深度模型在各种复杂的任务中表现十分优秀。例如卷积神经网络(CNN)这种由生物启发而诞生的网络,它基于数学的卷积运算而能检测大量的图像特征,因此可用于解决多种图像视觉应用、目标分类和语音识别等问题。但是,深层网络...
摘要:通过将神经元的值设置为希望的模式来训练网络,之后可以计算权重。输入神经元在完整网络更新结束时变成输出神经元。在某种程度上,这类似于峰值神经网络,并不是所有的神经元始终都在发射并且点的生物合理性得分。 随着新的神经网络架构不时出现,很难跟踪这些架构。知道所有缩写(DCIGN,BiLSTM,DCGAN,任何人?)起初可能有点压倒性。 所以我决定编写一个包含许多这些体系结构的备忘单。这些大多...
阅读 1157·2023-01-14 11:38
阅读 1149·2023-01-14 11:04
阅读 982·2023-01-14 10:48
阅读 2508·2023-01-14 10:34
阅读 1236·2023-01-14 10:24
阅读 1116·2023-01-14 10:18
阅读 733·2023-01-14 10:09
阅读 812·2023-01-14 10:02